|
这一期博主和大众一块学习一下癌症芯片表达数据关联的数据库。尽管日前转录组深度测序技术被广泛应用,基因芯片技术依靠其低成本和癌症科研的大样本量需求使得芯片数据日前仍在持续的产生。与RNA-seq数据产生的方式有所不同,芯片数据产生的平台多样,平台之间的数据常常不可直接比较。这针对芯片数据跨平台的比较和整合分析是有害的。幸运的是,有些很好的芯片数据库和分析平台被研发。
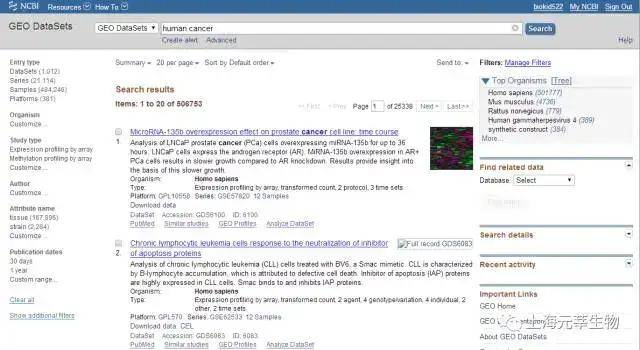
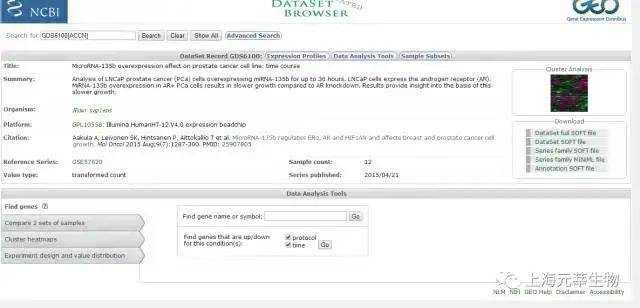
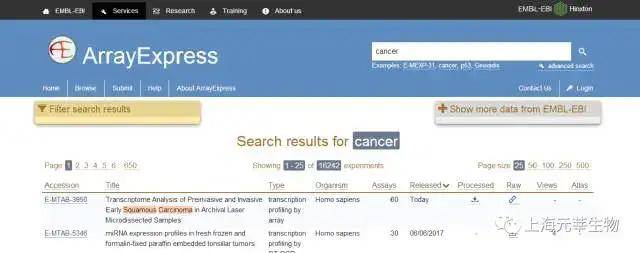
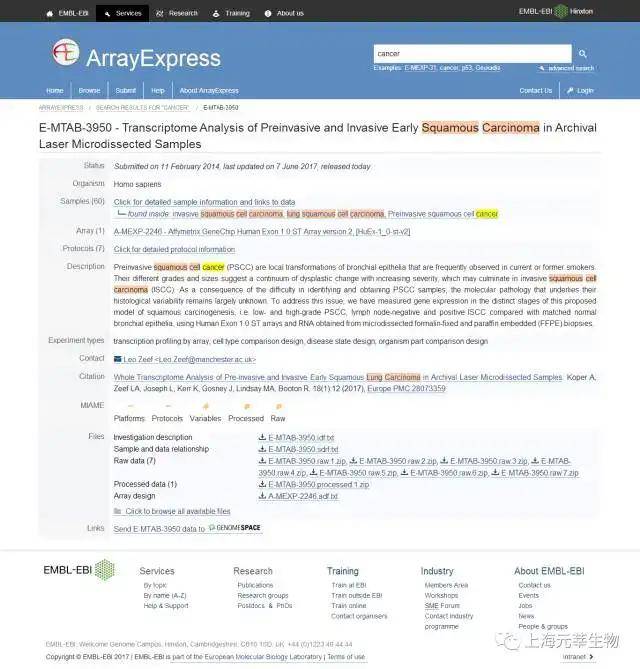
GEO (https://www.ncbi.nlm.nih.gov/geo/)与ArrayExpress(http://www.ebi.ac.uk/arrayexpress/)存储了大部分已公开的基因芯片表达原始数据和归一化数据,重点是源自于全世界专家的研究工作。当然,这亦包含海量的癌症科研工作。针对GEO,用户能够利用重要词进行搜索,如Humancancer。图1.1是搜索结果页面。用户能够经过左边栏选项对查找结果进行过滤。图1.2是一个数据集的下载页面。针对ArrayExpres,用户亦能够利用重要词进行搜索,如Cancer,图1.3是结果页面。一样用户亦能够进行数据过滤和数据下载(图1.4)【1-2】。
添加照片注释,不超过 140 字(可选)
图1.1 GEO 查找结果页面
添加照片注释,不超过 140 字(可选)
图1.2 GEO 数据集下载页面
添加照片注释,不超过 140 字(可选)
图1.3 ArrayExpress查找结果页面
添加照片注释,不超过 140 字(可选)
图1.4 ArrayExpress数据集下载页面
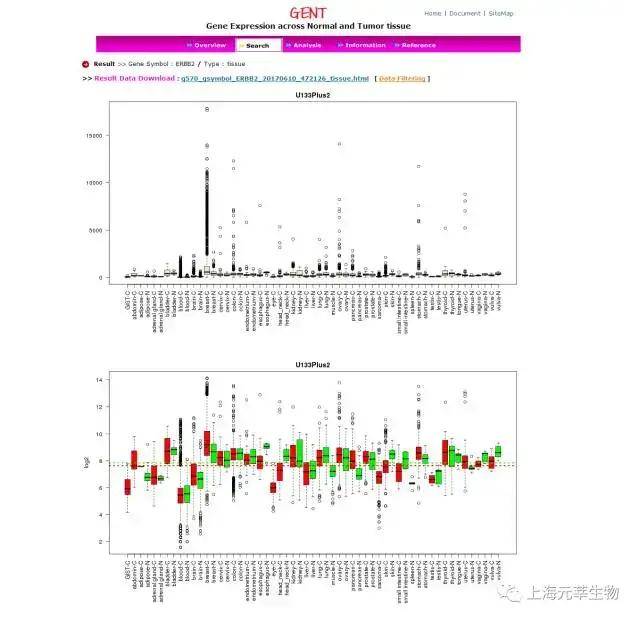
GENT(http://medicalgenome.kribb.re.kr/GENT/)收集了41000个癌症组织、癌症细胞系、和正常组织的芯片表达数据。并通图形化展示了在组织、细胞系中的表达模式。用户能够基于表达模式,找到有些表达反常的基因。用户能够按照基因名或Affymetrix ID搜索。图1.5展示了ERBB2在癌症和正常组织中表达状况。另外,用户还能够同期搜索多个基因在多个组织中的表达模式状况【3】。
添加照片注释,不超过 140 字(可选)
图1.5 ERBB2基因在癌症和正常组织中的表达
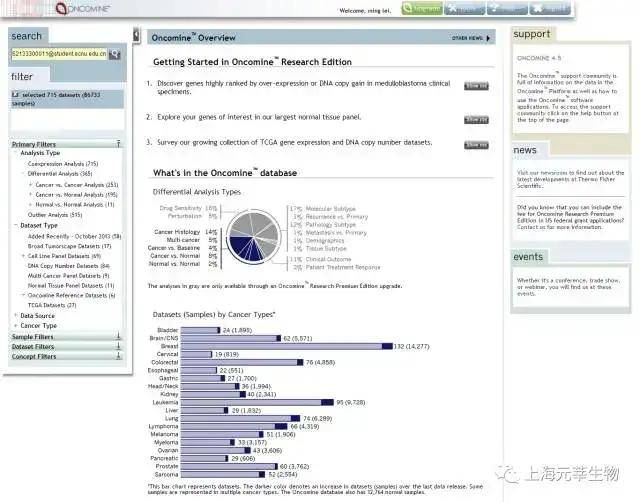
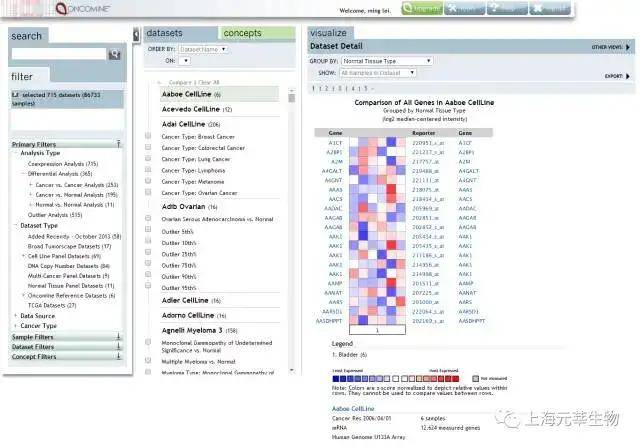
Oncomine(https://www.oncomine.org/resource/login.html)是一个癌症芯片数据库和整合型的数据挖掘平台。该数据库收集了715套芯片表达数据集,共包括了86733个样本以及关联的临床数据。用户既能够得到一个基因的差异表达分析,亦能够得到某一个科研工作中所有差异表达的基因。差异表达分析能够在癌症与正常组织之间、不同癌症亚型组织之间、基于临床和病理数据归类的样本之间进行。另外,用户还能够进行多基因搜索、依据Gene ontology和临床的注释信息进行过滤。图1.6是Oncomine主界面,图1.7是Oncomine数据呈现界面【4】。
添加照片注释,不超过 140 字(可选)
图1.6 Oncomine主界面
添加照片注释,不超过 140 字(可选)
图1.7 Oncomine数据呈现页面
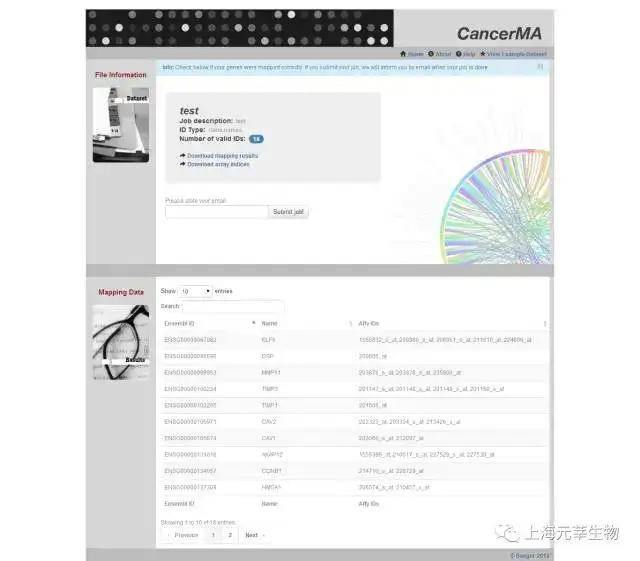
CancerMA(http://www.cancerma.org.uk/)一个芯片数据meta-analysis分析平台。其芯片数据源自于GEO与ArrayExpress。日前,包括了来自13种癌症的80套数据集。用户上传关心的基因名列表之后,该数据库会基于基因表达信息进行meta-analysis来找到新的候选癌症生物标志物。图1.8是数据最后的提交页面,用户需供给邮箱位置。图1.9是CAV1基因在肺癌中meta-analysis分析部分结果【5】。
添加图片注释,不超过 140 字(可选)
图1.8 CancerMA数据提交页面
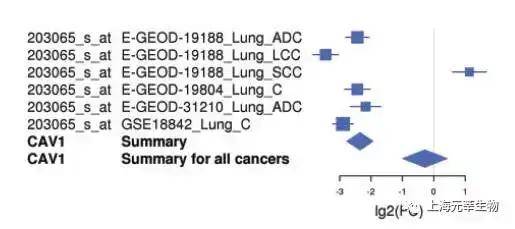
添加照片注释,不超过 140 字(可选)
图1.9 CAV1基因在肺癌中分析结果
参考文献
1. BarrettT, Edgar R: Gene expression omnibus: microarray data storage, submission,retrieval, and analysis. Methods in enzymology 2006, 411:352-369.
2. ParkinsonH: ArrayExpress--a public repository for microarray gene expression data at theEBI. Nucleic acids research 2004, 33(Database issue) 553-D55 553-D55
3. seon-YoungK, Gwangsik S, Kang, Sungjin Y, Su-Jin B, Yong-Su J: GENT: Gene ExpressionDatabase of Normal and Tumor Tissues. Cancer Informatics 2011:149.
4. RhodesDR, Yu J, Shanker K, Deshpande N, Varambally R, Ghosh D, Barrette T, Pandey A,Chinnaiyan AM: ONCOMINE: a cancer microarray database and integrateddata-mining platform. Neoplasia 2004, 6(1):1-6.
5. FeichtingerJ, McFarlane RJ, Larcombe LD: CancerMA: a web-based tool for automaticmeta-analysis of public cancer microarray data. Database : the journal ofbiological databases and curation 2012, 2012:bas055.返回外链论坛:http://www.fok120.com/,查看更加多
责任编辑:网友投稿
|