|
癌症科研始终是生物医学行业的热点。尤其是随着高通量基因测序技术的显现,癌症关联的生物数据呈现着指数式增长。这类数据繁杂而多样,包括了基因组变异、基因表达、甲基化数据等。因此呢,为了使更加多的研究人员能够方便有效地运用这些数据,越来越多的数据库与网页服务器工具被设计。本期博主就先和大众一块学习与癌症基因表达关联的数据库吧(表1.1)。
表1.1. 癌症基因表达数据库

添加照片注释,不超过 140 字(可选)
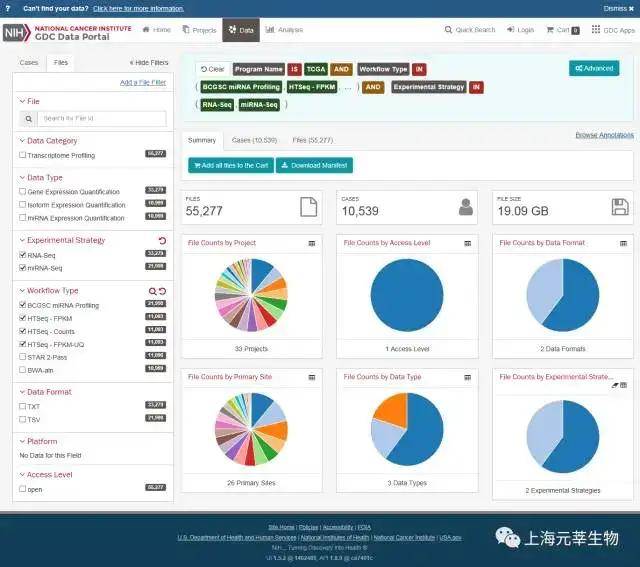
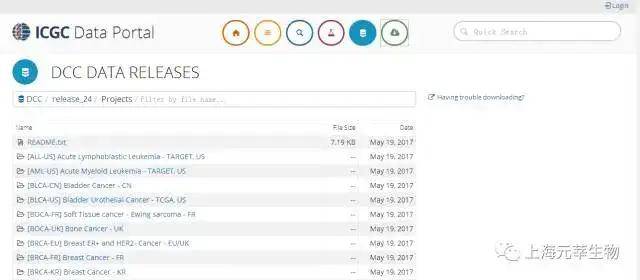
TCGA(https://cancergenome.nih.gov/)与ICGC(https://dcc.icgc.org/)都是综合型数据库。它们的数据都是源自于有些大规模癌症合作项目,数据信息最为丰富。然而无供给交互式的分析服务,相当于一级数据库。因此呢,这两个数据库只供给了基因表达数据(FPKM归一化或readscounts数据)的下载服务(图1.1,图1.2)。
添加照片注释,不超过 140 字(可选)
图1.1. TCGA RNA-seq数据
添加照片注释,不超过 140 字(可选)
图1.2. ICGC中数据下载页面
CGWB(https://cgwb.nci.nih.gov/)实质上是一个癌症数据可视化的数据库。其基因表达数据重点来自于TCGA。对这部分数据展示,CGWB供给了两种方式:1.以柱状图形式呈现每一个癌症样本中基因的表达水平分布(图1.3);2.以热图形式将该数据与其它数据(如拷贝数变异数据、临床数据)进行了整体展示。

添加照片注释,不超过 140 字(可选)
图1.3. TCGABLCA(Bladderurothelial carcinoma)病人的基因表达分布
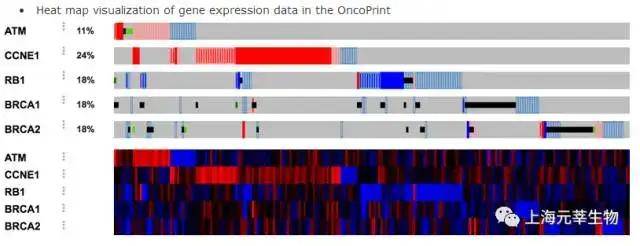
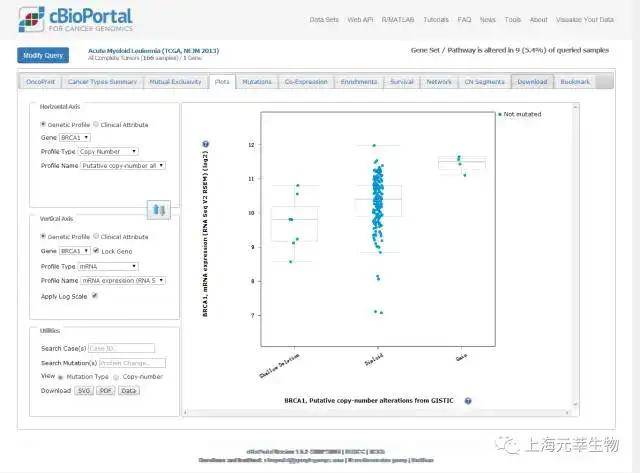
cBioPortal(http://www.cbioportal.org/)是一个综合性癌症数据分析数据库,供给了数据查找、展示、分析以及下载等功能。日前,该数据库收录了体细胞突变、DNA拷贝数变异、基因表达、DNA甲基化、蛋白质丰度和临床等数据。这些数据重点来自于TCGA、CCEL(CancerCell Line Encyclopedia)和有些癌症科研工作。咱们能够经过四步来进行数据的查找(如图1.4)。针对基因表达数据的分析重点包含了热图整体性展示(图1.5)、与其它数据(如拷贝数变化、DNA甲基化以及蛋白质水平)的相关分析(图1.6)、依据临床信息划分的样本集之间的表达比较分析(图1.6)、基因间表达关联性分析(图1.7)。

添加照片注释,不超过 140 字(可选)
图1.4.cBioPortal查找页面
添加照片注释,不超过 140 字(可选)
图1.5.基因表达热图
添加照片注释,不超过 140 字(可选)
图1.6. cBioPortal Plots界面
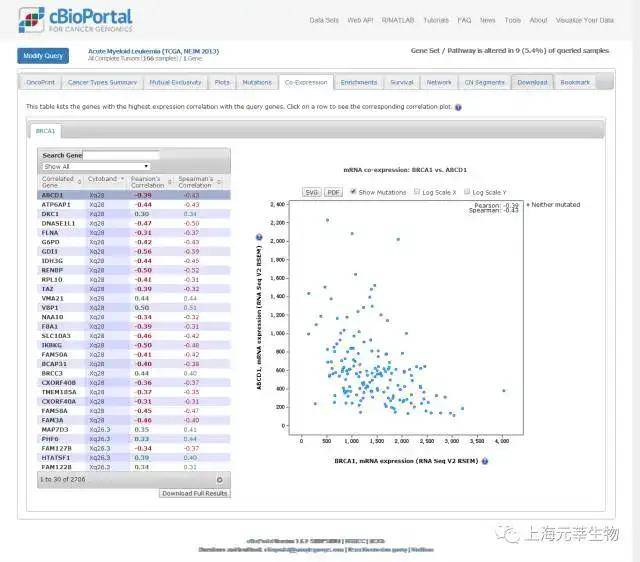
添加照片注释,不超过 140 字(可选)
图1.7. cBioPortalco-expression界面
GEPIA(http://gepia.cancer-pku.cn/index.html)是一个基因表达数据交互式分析的数据库,表达数据重点来自TCGA和GTEx(https://www.gtexportal.org/home/)。日前收录了9736个癌症组织样本(33种癌症)和8587正常组织样本的表达数据。该数据库功能非常强大,供给了差异表达分析、动态展示、基于基因表达的存活分析、表达类似基因分析、基因表达关联性以及主成份分析。并供给了非常丰富结果展示(图1.8)。

添加照片注释,不超过 140 字(可选)
图1.8. GEPIA分析的结果展示
CRN(http://syslab4.nchu.edu.tw/)数据库亦是一个基因表达数据分析数据库。其表达数据重点源自于GEO(https://www.ncbi.nlm.nih.gov/geo/)与TCGA。日前收录了28种癌症共11447个样本的表达数据,并按照样本的临床信息将每种癌症分成为了若干个子数据集。该数据库运用简单直接。咱们只需选取了癌症类型和配对子集,就能够进行差异表达分析与mRNA-lncRNA共表达网络构建(图1.9)。
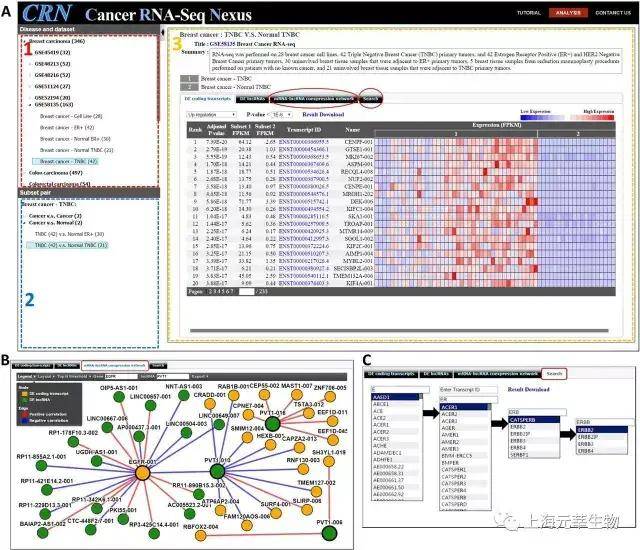
添加照片注释,不超过 140 字(可选)
图1.9. CRN数据库
tRF2Cancer(http://rna.sysu.edu.cn/tRFfinder/)是一个网页服务型数据库。供给了基于小RNA深度测序数据的tRFs(tRNA-derived small RNA Fragments)鉴定工具-tRFfinder;估计癌症样本中tRFs表达丰度工具-tRFinCancer以及基因组展示tRFs的工具-tRFBrowser。日前,该数据库共鉴定了TCGA中32种癌症共10991个样本的tRFs。咱们只需输入fasta格式的小RNA序列(图1.10),就能够得到预测的tRFs序列关联信息。包含序列、结构、表达丰度、基因组位置等信息(图1.11)。
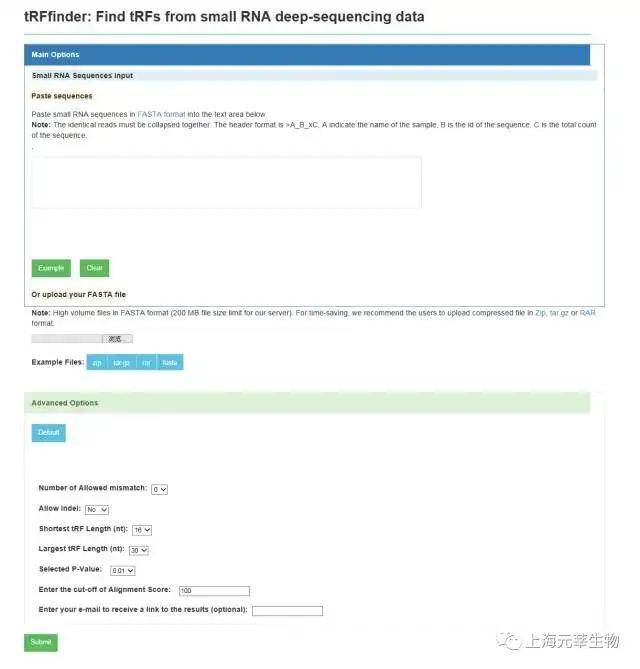
添加照片注释,不超过 140 字(可选)
图1.10. tRFfinder 提交页面
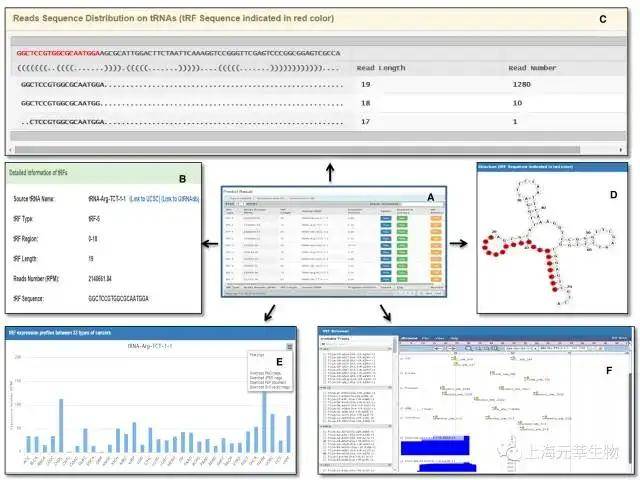
添加照片注释,不超过 140 字(可选)
图1.11. tRF2Cancer查找结果
dbDEMC 2.0 (http://www.picb.ac.cn/dbDEMC/)是一个存储和展示癌症样本中差异表达miRNA的数据库。日前,该数据库收录了36种癌症共2224个差异表达miRNA。这些基因是基于GEO和TCGA中209套数据集分析得到的。咱们能够基于基因信息或科研实验(experiments)来查找miRNA结果。如图1.12中A和C,点击差异基因列表中miRNA ID能够得到这个基因的仔细信息(E)。另外,该数据库还能够经过选取多个癌症,用热图的形式展示了miRNA的差异表达信息(图1.13)。

添加照片注释,不超过 140 字(可选)
图1.12. dbDEMC 2.0数据库
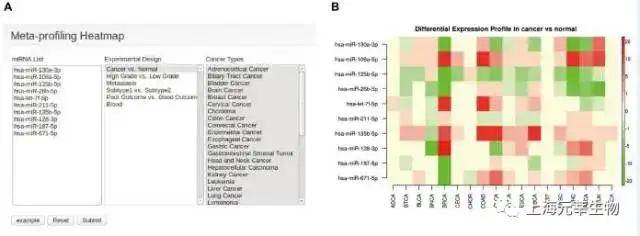
添加照片注释,不超过 140 字(可选)
图1.13. dbDEMC 2.0数据库的Meta-profiling Heatmap分析
ISOexpresso(http://wiki.tgilab.org/ISOexpresso/)是一个供给癌症样本中转录本表达信息和分析的数据库。该数据库日前收录了TCGA中30中癌症类型共10422样本的基因和转录本表达信息。咱们能够经过选取不同组织、癌症类型和基因名进行搜索(图1.14a)。查找结果包括了该基因转录本的注释信息以及不同转录本之间的表达状况(图1.14 b)。倘若咱们同期选取了癌症和正常样本(Normal-tumor comaprison选项),数据库还会给出转录本肿瘤特异性信息(图1.15)。另一,我们还能够上传癌症突变数据,该数据库会基于该癌症中转录本表达信息,对突变数据进行注释。

添加照片注释,不超过 140 字(可选)
图1.14. ISOexpresso数据库查找和结果呈现
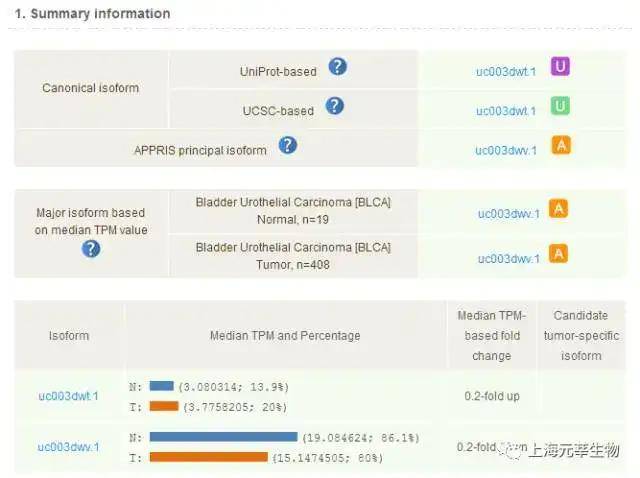
添加照片注释,不超过 140 字(可选)
图1.15. 癌症组织特异性信息
以上便是这期的所有内容啦,博主相信这些数据库必定会给咱们带来意想不到的方便。还有,这儿介绍的是RNA-seq数据的癌症数据库哦,还有非常多基于基因芯片数据的癌症数据库。那就下期再会了。
参考文献
1. Cancer Genome Atlas Research N, WeinsteinJN, Collisson EA, Mills GB, Shaw KR, Ozenberger BA, Ellrott K, Shmulevich I,Sander C, Stuart JM: The Cancer Genome Atlas Pan-Cancer analysis project.Nature genetics 2013, 45(10):1113-1120.
2. International Cancer Genome C, Hudson TJ,Anderson W, Artez A, Barker AD, Bell C, Bernabe RR, Bhan MK, Calvo F, Eerola Iet al: International network of cancer genome projects. Nature 2010,464(7291):993-998.
3. Zhang J, Finney RP, Rowe W, Edmonson M, YangSH, Dracheva T, Jen J, Struewing JP, Buetow KH: Systematic analysis of geneticalterations in tumors using Cancer Genome WorkBench (CGWB). Genome research2007, 17(7):1111-1117.
4. Gao J, Aksoy BA, Dogrusoz U, Dresdner G,Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E et al: Integrativeanalysis of complex cancer genomics and clinical profiles using the cBioPortal.Science signaling 2013, 6(269):pl1.
5. Tang Z, Li C, Kang B, Gao G, Li C, Zhang Z:GEPIA: a web server for cancer and normal gene expression profiling andinteractive analyses. Nucleic acids research 2017.
6. Li JR, Sun CH, Li W, Chao RF, Huang CC, ZhouXJ, Liu CC: Cancer RNA-Seq Nexus: a database of phenotype-specifictranscriptome profiling in cancer cells. Nucleic acids research 2016,44(D1) 944-951. 944-951.
7. Zheng LL, Xu WL, Liu S, Sun WJ, Li JH, Wu J,Yang JH, Qu LH: tRF2Cancer: A web server to detect tRNA-derived small RNAfragments (tRFs) and their expression in multiple cancers. Nucleic acidsresearch 2016, 44(W1):W185-193.
8. Yang Z, Wu L, Wang A, Tang W, Zhao Y, ZhaoH, Teschendorff AE: dbDEMC 2.0: updated database of differentially expressedmiRNAs in human cancers. Nucleic acids research 2017, 45(D1) 812-D818. 812-D818.
9. Yang IS, Son H, Kim S, Kim S: ISOexpresso: aweb-based platform for isoform-level expression analysis in human cancer. BMCgenomics 2016, 17(1):631.返回外链论坛:http://www.fok120.com/,查看更加多
责任编辑:网友投稿
|